In kaart brengen van verspreiding van antibacteriële resistentie in en rond het ziekenhuis met long-read sequencing
22-2-2021Antimicrobiële resistentie ligt vaak niet gecodeerd in het bacteriële chromosomale DNA, maar op aparte, mobiele genetische elementen zoals plasmiden en transposons, die los van het chromosoom vermenigvuldigd worden. Deze elementen kunnen terechtkomen in andere soort bacteriën. Met nieuwe DNA-sequencingtechnieken (long-read sequencing) kunnen deze mobiele genetische elementen nauwkeurig in kaart gebracht worden en kan de verspreiding van antimicrobiële resistentie worden onderzocht.
Onderzoekers Casper Jamin en Lieke van Alphen van het MUMC houden zich bezig met het in kaart brengen van de verspreiding van antimicrobiële resistentie bij bacteriën in en rond het ziekenhuis.
Antibioticaresistente bacteriën vormen een groot gevaar voor patiënten in het ziekenhuis. Verspreiding van deze bacteriën moet dan ook zoveel mogelijk voorkomen worden. Bij verdenking van een uitbraak worden de bacteriële stammen getypeerd, waarmee bepaald kan worden of ze onderdeel zijn van een uitbraakcluster. Klassieke typeringsmethodes worden langzaam vervangen door Whole genome sequencing (WGS). Hiermee kan het hele bacteriële genoom in kaart gebracht worden. Naast dat bacteriën zich kunnen verspreiden, kunnen ze ook hun antibioticaresistentie doorgeven aan andere bacteriën. Dit gebeurt door de conjugatie van plasmiden. Met long-read sequencing kunnen ook losse plasmiden als individuele units in het genoom herkend worden.
Plasmiden zijn stukken DNA die los van het chromosoom repliceren. Vaak hebben ze genen bij zich die coderen voor antibioticaresistentie. De orde Enterobacterales, waar onder andere Escherichia coli, Klebsiella en Enterobacter onder vallen, zorgt vaak voor problemen in het ziekenhuis. Bacteriën van deze groep kunnen plasmiden met elkaar uitwisselen waardoor resistentie tegen antibiotica wordt doorgegeven.
Voor de detectie van bacteriële uitbraken in het ziekenhuis wordt gekeken naar de mutaties die ontstaan in geconserveerde genen in het genoom. Plasmiden zijn echter veel minder geconserveerd en integreren vaak ook nieuwe stukken DNA zoals transposons en integrons.
Om toch inzicht te krijgen of er mogelijk uitbraken zijn waarbij plasmiden betrokken zijn, zijn er nieuwe analysemethoden nodig om te zien of dezelfde plasmiden gevonden worden bij verschillende bacteriën. Door de integratie van nieuwe stukken DNA is het niet gemakkelijk om de sequenties direct te vergelijken. Door te kijken naar welke sub-sequenties van lengte k (k-mer) (figuur 1) voorkomen tussen twee plasmiden, kan de similariteit-index tussen deze twee DNA-sequenties berekend worden.
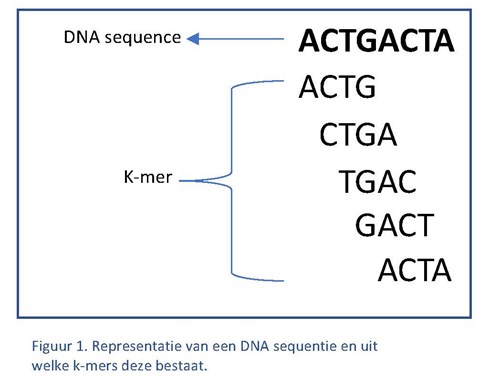
Op basis van deze similariteit-index kunnen verschillende plasmiden gegroepeerd worden. Hiervoor heeft Casper Jamin een analyse-pijplijn genaamd ‘Plasmidsimilarity’ ontwikkeld. Hiermee worden eerst resistentie-en virulentie-genen op de plasmiden in kaart gebracht en vervolgens de DNA-sequenties van de plasmiden geclusterd.
source code Plasmidsimilarity
Onderzoekers:
Casper Jamin, Promovendus Medische Microbiologie, Maastricht UMC+ (casper.jamin@mumc.nl)
- Lieke van Alphen, Medisch Moleculair Microbioloog, Maastricht UMC+ (lieke.van.alphen@mumc.nl)