Samenvatting
In het huidige Rijksvaccinatieprogramma wordt vaccinatie tegen bof, mazelen en rodehond aangeboden aan alle kinderen op de leeftijd van 14 maanden en 9 jaar. Infecties met voornamelijk bof- en mazelenvirus komen in Nederland (en vele andere landen) echter nog steeds voor. Grote uitbraken van bof vonden plaats tussen 2009 en 2012. De laatste grote uitbraak van mazelen was in 2013-2014 en van rubella in 2004-2005. Jaarlijks zijn er geregeld sporadische gevallen en kleine clusters/uitbraken van mazelen- en bofvirus. Door het systematisch verzamelen van gegevens over het vóórkomen van infecties met deze virussen wordt in kaart gebracht waar en hoe vaak deze ziekten nog voorkomen en kunnen uitbraken gesignaleerd worden. Moleculaire surveillance speelt hier een belangrijke rol in. Hiertoe worden sequenties bepaald van virussen die in verschillende laboratoria zijn gedetecteerd. Door het onderzoeken van (vrijwel) complete genomen van virussen kan de moleculaire resolutie vergroot worden, waardoor op een fijner niveau clustering kan worden aangetoond. Nieuwe sequentietechnieken spelen hier een belangrijke rol in, waarbij de inzet van deze technieken op globale schaal nodig is om de meest bruikbare informatie te verkrijgen.
Abstract
According to the current national immunization program in the Netherlands, vaccination against mumps, measles and rubella is offered to all children aged 14 months and 9 years. However, infections with mainly mumps and measles virus still occur in the Netherlands (and many other countries). Major outbreaks of mumps occurred from 2009 to 2012, the last major outbreak of measles was in 2013-2014, and of rubella in 2004-2005. In addition, there are occasional sporadic cases and small clusters/outbreaks of measles and mumps. By systematically collecting data on the occurrence of infections with these viruses, it is mapped where and how often these diseases still occur and outbreaks can be identified. Molecular surveillance plays an important role in this. For this purpose, sequences are determined of viruses detected in different laboratories. For measles and rubella virus, online (closed) databases have been set up by the WHO to which sequences are added worldwide. Molecular resolution can be increased by determining (near) complete genomes of viruses. New sequencing techniques play an important role in this and the use of these techniques on a global scale is crucial to obtain the most useful information.
Introductie
Bof, mazelen en rodehond zijn virale aandoeningen, veroorzaakt door drie verschillende virussen. Sinds 1987 is er in Nederland een gecombineerd vaccin beschikbaar waarin levende verzwakte varianten van deze drie virussen zitten. Dit vaccin wordt volgens het huidige schema van het Rijksvaccinatieprogramma aangeboden aan kinderen van 14 maanden en van 9 jaar.
Zowel het bof-, mazelen- als rodehond virus zijn RNA-virussen, maar behoren tot verschillende families. Het bofvirus en mazelenvirus behoren tot de familie van de Paramyxoviridae, terwijl het rodehondvirus tot de familie van de Togaviridae behoort. Genetisch zijn de drie virussen relatief stabiel. Het is dan ook niet nodig dat deze virussen in het vaccin regelmatig aangepast worden aan op dat moment circulerende virusstammen. De levend verzwakte bof-, mazelen- en rodehondvirusvaccinstammen die enkele decennia geleden zijn ontwikkeld, zijn nog steeds in staat om voldoende immuniteit te induceren tegen de virussen die op dit moment circuleren.[1,2]
In het Global Vaccine Action Plan 2011-2020 van de Wereldgezondheidsorganisatie (WHO) is besloten om te streven naar eliminatie van mazelen en rubella in verschillende delen van de wereld. In Nederland komt mazelen buiten de grote uitbraken onder niet-gevaccineerden weinig meer voor, maar wereldwijd overleden er in 2017 nog meer dan 100.000, voornamelijk kinderen.[3] Om de wereldwijde verspreiding van mazelenvirus te stoppen wordt een tweedosisschema aanbevolen, met een vaccinatiegraad van minimaal 95 procent voor beide doses.[1]
Om de impact van het vaccinatieprogramma te evalueren is, naast het bepalen van de vaccinatiegraad, surveillance nodig. De surveillance bestaat uit ziekte- en laboratoriumsurveillance. Door middel van het analyseren van sequentiedata (moleculaire surveillance) van pathogenen kunnen gedetecteerde uitbraken verder gekarakteriseerd en transmissieketens geïdentificeerd worden. Moleculaire surveillance kan laten zien of er in een bepaald land of gebied sprake is van endemische circulatie van een virus of dat er alleen sprake is van importgevallen. In de Nederlandse situatie – met een relatief hoge vaccinatiegraad en lage incidentie - is het belangrijk om van elk sporadisch geval en van minimaal een deel van de gevallen in een nieuwe transmissieketen materiaal te verzamelen voor moleculaire surveillance.
In dit artikel bespreken we de achtergrond van de moleculaire surveillance van bof-, mazelen- en rodehondvirus. Daarnaast geven we een overzicht van de resultaten van de moleculaire surveillance van bof-, mazelen en rodehondvirus in Nederland van de afgelopen jaren. Ook worden op basis van recente data de mogelijkheden en beperkingen van moleculaire surveillance aangegeven.
Surveillance
Bof, mazelen en rodehond zijn meldingsplichtige infectieziekten. In het kader van de Wet publieke gezondheid ontvangen GGD’s meldingen van medisch-microbiologische laboratoria en artsen. De GGD voert bron- en contactonderzoek uit en verzamelt aanvullende informatie over onder andere eventuele complicaties, de vaccinatiestatus en de meest waarschijnlijke herkomst van besmetting. Deze gegevens worden met beperkte persoonsgegevens (geslacht, geboortejaar, viercijferige postcode) geregistreerd in Osiris, een onlinesysteem dat beheerd wordt door het RIVM. Met de gegevens uit deze ziektesurveillance wordt de epidemiologie beschreven en wordt onderzocht welke risicofactoren een rol kunnen spelen en welke interventies effectief zijn. Naast de ziektesurveillance wordt materiaal opgevraagd voor typering. Moleculaire surveillance kan informatie geven over de bron en transmissiepatronen. Daarnaast is moleculaire surveillance belangrijk voor de certificering van eliminatie.
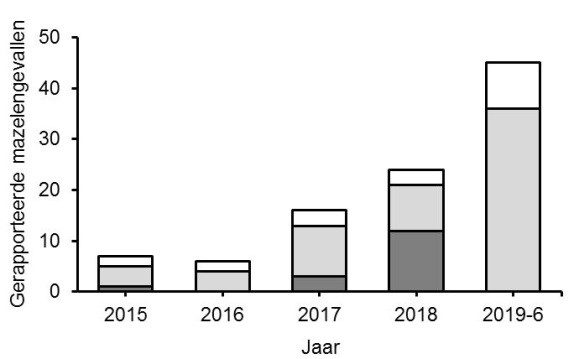
De laatste grote uitbraak van mazelen was in 2013-2014, van bof waren er in de periode 2009 tot 2012 achtereenvolgende uitbraken onder studenten (over drie seizoenen), en de laatste grote uitbraak van rubella was in 2004-2005.[4-6] In de periode 2015 tot en met juni 2019 was de incidentie van mazelen minder dan 1 per miljoen inwoners in 2015-2017, 1,4 in 2018 en 5,2 per miljoen in de eerste helft van 2019. Ter vergelijking was de incidentie 160 gevallen per miljoen tijdens de laatste epidemie in 2013. De incidentie van bof varieerde in deze periode tussen 2,7 (2017) en 7,2 per miljoen (eerste helft 2019). Van rubella werd sinds 2015 slechts één geval gemeld.
Endemische transmissie binnen een bepaald gebied is volgens de WHO gedefinieerd als de continue transmissie van een inheems of geïmporteerd mazelen- of rubellavirus gedurende 12 of meer maanden.[7] In Nederland is dat de afgelopen 36 maanden niet aan de orde geweest. Zelfs bij de laatste uitbraak van mazelen in 2013-2014 was het virus binnen 12 maanden geëlimineerd, wat we te danken hebben aan de algeheel hoge graad van immuniteit in de Nederlandse bevolking.
Moleculaire surveillance
Moleculaire surveillance van het bof-, mazelen- en rodehondvirus is op dit moment gebaseerd op de sequentie van beperkte delen van de genomen van deze virussen. De te onderzoeken delen van het genoom zijn in eerste instantie geselecteerd op hun relatieve grote genetische variatie ten opzichte van andere delen van het genoom. Aangezien wereldwijd dezelfde delen van het genoom onderzocht worden, kunnen relatief goede vergelijkingen gemaakt worden.
Mazelenvirus
Voor de genotypering van het mazelenvirus (15.894 baseparen) beveelt de WHO aan om minimaal de sequentie te karakteriseren van de 450 nucleotiden (N-450) coderend voor de 150 aminozuren van het COOH-einde van het kerneiwit (N). De WHO heeft acht verschillende groepen onderscheiden op basis van genetische verschillen, de groepen A tot en met H. Binnen die groepen worden er op dit moment 24 genotypen onderscheiden, te weten A, B1-3, C1, C2, D1-11, E, F, G1-3 en H1-2.[8] Alle vaccinstammen behoren tot genotype A. Er zijn geen aanwijzingen dat er nog genotype A-wildtype-mazelenvirussen circuleren. In het geval dat genotype A wordt gedetecteerd, bijvoorbeeld tijdens uitbraken, is er sprake geweest van een kort daarvoor uitgevoerde mazelenvaccinatie.
Volgens de richtlijnen van de WHO worden alle door WHO-referentielaboratoria verzamelde sequenties van gedetecteerde mazelenvirussen verstuurd naar de online database Measles Nucleotide Surveillance (MeaNS; www.who-measles.org). Op deze manier ontstaat inzicht in de wereldwijde verspreiding van genetische varianten van mazelenvirussen, al is er sprake van een duidelijke bias aangezien niet in alle landen sequenties worden gekarakteriseerd en/of ingestuurd van gedetecteerde mazelenvirussen.[9]
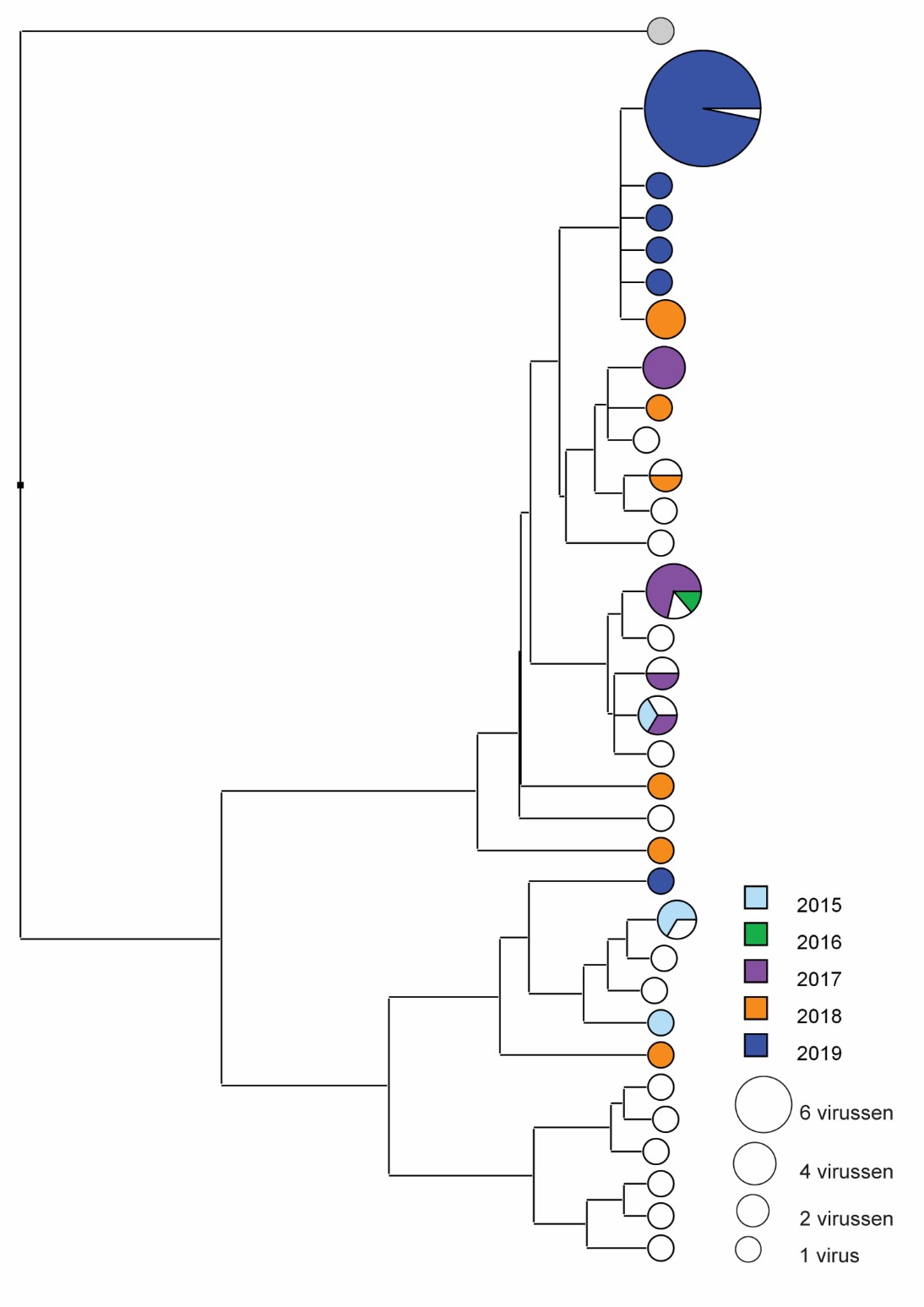
In tabel 1 wordt een overzicht gegeven van het aantal meldingen van mazelen in Nederland per jaar in de periode 2015 tot en met juni 2019. De patiënten waren tussen de 0 en 57 jaar (mediaan 28). Mazelenvirussen van het genotype B3 en D8 veroorzaken de recente grote uitbraken van mazelen in Europa. Ook de mazelenuitbraak in Nederland in 2013-2014 werd veroorzaakt door het D8-genotype. In de jaren daarna werden in Nederland ook alleen infecties met deze genotypen gerapporteerd. In de periode 2015 tot en met juni 2019 was van 84 van de 98 patiënten een monster beschikbaar voor typering. In 79 gevallen (81 procent) kon ook daadwerkelijk het genotype bepaald worden, 60 daarvan behoorden tot het genotype D8 (figuur 1). Er werden in deze periode 17 epidemiologische clusters gemeld van tussen de twee en elf patiënten.[11] In totaal was 61 procent van de patiënten onderdeel van een cluster, dit aandeel was hoger in de jaren met een groter aantal meldingen (tabel 1). Het merendeel van de patiënten (68 procent) heeft de infectie in het buitenland opgelopen of was gerelateerd aan een importgeval. Van 14 clusters was er van ten minste twee patiënten materiaal voor typering beschikbaar. Vijf clusters waren op basis van een unieke D8-sequentie ook als een apart cluster te onderscheiden, de andere negen clusters waren moleculair niet van elkaar te onderscheiden. De sequenties van 29 van de 36 mazelenvirussen, waarvan in 2019 een genotype gekarakteriseerd kon worden, waren identiek (figuur 2). Dit dendrogram is gemaakt op basis van UPGMA (unweighted pair group method with arithmetic mean) en bevat 60 in Nederland gedetecteerde genotype D8- mazelenvirussen in 2015-2019 (met kleur weergegeven) en 20 epidemiologisch relevante genotype D8- sequentievarianten van mazelenvirus de afgelopen jaren (‘named strains’, wereldwijd; met wit weergegeven). De grootte van de cirkels geeft het aantal virussen weer en de kleuren van de (parten van de) cirkels de jaren waarin die virussen gedetecteerd zijn. Het in grijs weergegeven virus is een genotype D1-mazelenvirus; MVi/Bristol.GBR/0.74, wat gebruikt is als referentiestam voor het bepalen van de phylogenetische afstand tussen de virussen (outgroup). De dendrogram is gemaakt met BioNumerics versie 7.6.3
Uit de data van de moleculaire surveillance van mazelenvirus in Nederland in 2019 valt niet of zeer beperkt op te maken of er sprake is van circulatie van mazelenvirus in Nederland, of van verschillende importgevallen met dezelfde genotypesequentie (figuur 2). De waarde van moleculaire surveillance op basis van een relatief klein deel van het genoom is daarmee te beperkt wanneer uitbraken worden veroorzaakt door één genotypevariant, zoals in 2019 het geval is. Wanneer zo’n genotype bovendien domineert binnen heel Europa kunnen mazelenvirussen die in Nederland worden gedetecteerd bij importgevallen uit verschillende landen toch exact dezelfde sequentie hebben, zonder epidemiologische link. In dit geval kan de resolutie van de moleculaire surveillance vergroot worden door additionele onderdelen van het genoom of zelfs het complete genoom van mazelenvirussen te karakteriseren.[12-14] Op dit moment worden ook op in Nederland gedetecteerde mazelenvirussen verschillende methoden toegepast om sequenties van additionele delen van het genoom te verzamelen om zo de moleculaire resolutie te vergroten. De eerste resultaten geven al aan dat mazelenvirussen van verschillende recente epidemiologische clusters in Nederland door middel van het bepalen van additionele delen van het mazelenvirusgenoom ook moleculair van elkaar te onderscheiden zijn.
Rodehondvirus
Net als voor het mazelenvirus is voor rubellavirus (rodehond) een specifiek deel van het genoom geselecteerd voor de genotypering, in dit geval de 739 nucleotiden van het E1-gen (posities 8731-9469). Er zijn op dit moment 13 genotypen bekend, 1a, 1B-1J, 2A-C, waarbij 1a een zogeheten ‘provisional’ genotype is, waartoe het rubellavaccin behoort, maar waarvan wordt aangenomen dat deze als wildtypevirus niet meer circuleert.[15,16] Momenteel circuleren vooral nog genotype 1E en 2B wereldwijd. De laatste kleine uitbraak van rubellavirus van het type 2B was in Nederland in 2013, met meer dan 50 gevallen. In de jaren daarna zijn er in Nederland slechts enkele gevallen van rubellavirus gerapporteerd.[10]
Bofvirus
Voor de genotypering van bofvirussen wordt de complete nucleotidensequentie van het zogeheten small hydrophobic (SH) eiwit en de naast gelegen niet-coderende regio’s gebruikt. Dit is het meest variabele deel van het bofvirusgenoom.[17] In totaal zijn dit 316 nucleotiden. Er zijn verschillende primersets die gebruikt kunnen worden voor de genotyperingen van bofvirus. De WHO heeft enige jaren geleden voorgesteld om de gebruikte nomenclatuur voor de genotyperingen van bofvirussen aan te passen. Volgens de nieuwe nomenclatuur worden er nu 12 verschillende genotypen herkend, genaamd A-N met uitzondering van E, M. Daarnaast is er ook voorgesteld om genotype G-bofvirussen verder op te splitsen, gezien de genetische variabiliteit.[17]
Tussen 2009 en 2012 waren er grote uitbraken van bofvirus onder studenten.[5] Deze uitbraken werden vrijwel uitsluitend veroorzaakt door bofvirussen van het genotype G. Ook in de jaren daarna werden bofvirussen van het genotype G het meest gedetecteerd in Nederland. Zo nu en dan zijn er de afgelopen jaren bofvirussen met een ander genotype (C, D, H, K) gedetecteerd, als gevolg van import uit landen waar die genotypen circuleren.[17]
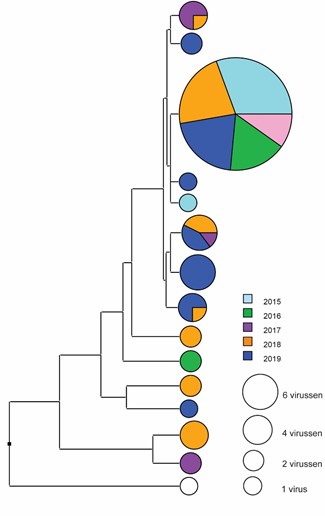
Hoewel het SH-gen voor het bofvirus het gebied is met de meeste genetische variatie, is ook deze regio genetisch relatief stabiel. Zo nu en dan zijn er importgevallen van genotype G-bofvirussen uit verschillende genetische clusters, maar het merendeel van de genotype G-bofvirussen die van 2014 tot en met 2019 in Nederland werden gedetecteerd zijn genetisch identiek. Dit suggereert dat in Europa/Nederland al enkele jaren een beperkt aantal bofvirussen circuleert, zie ook het dendrogram in figuur 3. Dit is gemaakt op basis van UPGMA en bevat 111 in Nederland gedetecteerde genotype G-bofvirussen in 2014-2019. De grootte van de cirkels geeft het aantal virussen weer en de kleuren van de (parten van de) cirkels de jaren waarin die virussen gedetecteerd zijn. Het virus weergegeven met de witte cirkel is de referentiestam MuVi/London.GBR/03.02. De dendrogram is gemaakt met BioNumerics versie 7.6.3.
In 2018 en de eerste zes maanden van 2019 waren er negen epidemiologische clusters bestaande uit 2 tot 14 patiënten. Van zes clusters van genotype G-bofvirus was meer dan één monster beschikbaar voor genotypering (totaal 19 monsters). Van bofvirussen gedetecteerd in vier (A-D) van deze clusters waren de SH-sequenties identiek (totaal 13 monsters).
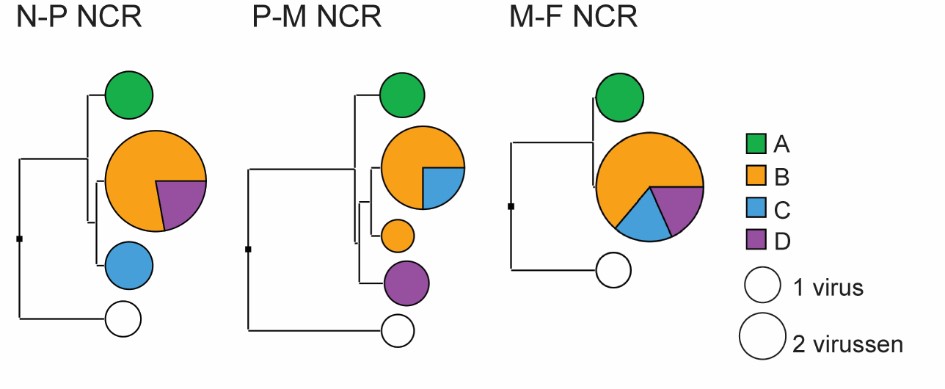
Net als bij het mazelenvirus, kan de resolutie van de moleculaire surveillance van bofvirussen vergroot worden door genoominformatie van aanvullende genen en/of regio’s te karakteriseren. Hiervoor kunnen bijvoorbeeld bepaalde niet-coderende gebieden (NCR) tussen het N- en P-gen, het P- en M-gen en het M- en F-gen (N-P, P-M, M-F NCR’s) of de genen coderend voor het HN- en F-eiwit worden gebruikt.[18-20] Uit analyse van de NCR-sequentiedata van de bofvirussen gedetecteerd in de vier clusters met identieke SH-sequentie in 2018-2019, bleek dat er sprake was van enige variatie waardoor virussen uit de verschillende clusters toch genetisch van elkaar onderscheiden kunnen worden (figuur 4). Recent was er in een grote stad sprake van een verhoogd aantal bofgevallen. Hoewel een gemeenschappelijke bron van het bofvirus hier het meest waarschijnlijk leek, bleek op grond van de sequentieanalyse dat er ten minste twee onderscheidende virusvarianten circuleerden. Dit laat zien dat we hier te maken moeten hebben gehad met ten minste twee transmissieketens van het bofvirus.
Toekomst moleculaire typering bof-, mazelen- en rodehondvirus
Door de ontwikkeling van nieuwe sequentietechnieken is het eenvoudiger geworden om aanvullende genetische informatie van virale genomen te verzamelen. Hierdoor is het mogelijk de moleculaire resolutie te vergroten en kunnen clusters en individuele gevallen nu beter aan elkaar gelinkt worden of juist worden herkend als afzonderlijke (import)gevallen. Op dit moment zijn er echter toch nog enkele uitdagingen om uit verschillende soorten monsters een betrouwbaar compleet viraal genoom te onderzoeken. Het voorbeeld van bofvirus en de huidige situatie van mazelenvirus in Nederland en Europa geven duidelijk de toegevoegde waarde van het verkrijgen van complete virale genomen aan. Daarnaast is het voor de moleculaire surveillance van mazelen- en bofvirus van groot belang dat in meerdere landen additionele genoominformatie wordt verzameld en dat deze data wordt gedeeld.
Dankwoord
Graag willen wij alle medewerkers van laboratoria en GGD’s bedanken voor het insturen van monsters voor genotyperingen. Daarnaast willen we de collega’s van het RIVM bedanken die betrokken zijn geweest bij het karakteriseren van de sequenties van de mazelen- en bofvirussen die gebruikt zijn voor de analyses in dit artikel.